Récapitulatif
Dans cette mise à jour, l'équipe de recherche Mapping Cancer Markers explique comment un changement de code relativement petit pourrait avoir un impact important sur la capacité du projet à analyser les marqueurs pour différents types de sarcome.
Contexte
Mapping Cancer Markers vise à identifier les marqueurs (parfois appelés signatures) associés à divers types de cancer. Le projet analyse des millions de points de données collectés à partir de milliers d'échantillons de tissus de patients sains et cancéreux. Jusqu'à présent, ceux-ci comprenaient des tissus atteints de cancer du poumon, de cancer de l'ovaire et de sarcome.
Élargir notre capacité à travailler avec des ensembles de données
Des changements de code arrivent bientôt pour améliorer le projet Mapping Cancer Markers. Ces changements élargiront la capacité de l'application Mapping Cancer Markers à traiter des ensembles de données multi-étiquettes et lui permettront de rechercher des signatures plus spécifiques en leur sein.
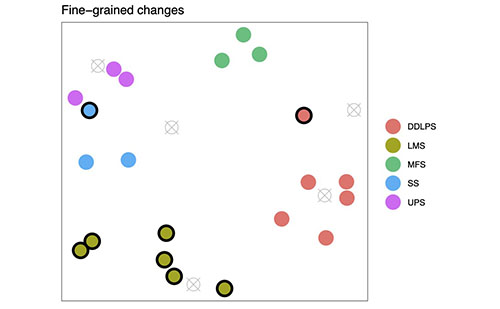
Figure 1A (ci-dessus) : Cela représente l'ensemble de données sur le sarcome, avec sept sous-types d'échantillons. Mapping Cancer Markers peut utiliser le jeu de données multi-étiqueté tel quel et rechercher des signatures multiclasses (un contre tous). (Le logiciel du projet peut le faire maintenant.)
Avant le jeu de données sur le sarcome, le projet a analysé des jeux de données sur le cancer du poumon et de l'ovaire, qui ont tous deux des étiquettes binaires. Notre ensemble de données sur le cancer du poumon a étiqueté les échantillons comme «cancer» ou «pas de cancer». Notre ensemble de données ovariennes a qualifié les échantillons de survie courte ou longue. L'ensemble de données sur le sarcome est multi-marqueur et marque les échantillons avec sept sous-types différents de sarcome (voir la figure 1A ci-dessus).
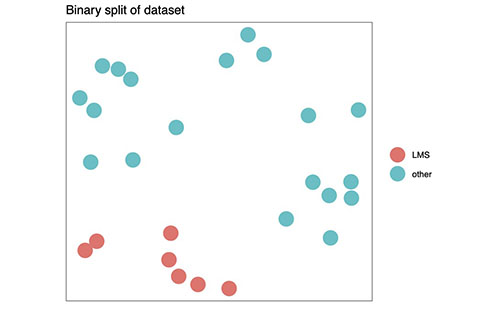
La figure 1B (ci-dessus) montre comment MCM peut réduire le sarcome à un ensemble de données binaires, en divisant les sous-types en deux groupes et en recherchant des signatures binaires. (Le logiciel du projet peut le faire maintenant.)

La figure 1C montre une réduction alternative à un ensemble de données binaires. (Le logiciel du projet peut le faire maintenant.)
Lorsqu'un ensemble de données a des étiquettes binaires, Mapping Cancer Markers trouvera des signatures qui peuvent prédire cette étiquette binaire. Avec un ensemble de données multi-étiquettes, cependant, nous pouvons diriger Mapping Cancer Markers pour rechercher des signatures binaires ou multiclasses (voir les figures 1B et 1C ci-dessus pour des exemples). Actuellement, le projet recherche les deux dans l'ensemble de données sur le sarcome. Une signature de sarcome multiclasse distingue chaque sous-type de tout autre; étant donné n'importe quel échantillon de sarcome, il diagnostiquera le sous-type de cancer spécifique. Une signature de sarcome binaire distingue un groupe de sous-types du reste, mais ne distingue pas les sous-types spécifiques. Par exemple, parmi les sept sous-types de sarcome, il y a deux sous-types de leimyosarcome (LMS), les tissus mous et l'utérus. Mapping Cancer Markers recherche actuellement des signatures binaires qui distinguent LMS des autres.

La figure 1D montre comment les nouvelles capacités de Mapping Cancer Marker permettront à une unité de travail de se concentrer sur des sous-types spécifiques. Les échantillons exclus sont gris et barrés. (Nouveau code.)

La figure 1E montre les nouvelles capacités du Mapping Cancer Marker dans leur intégralité, en excluant les échantillons individuels et en modifiant leurs étiquettes. Les échantillons réétiquetés sont encadrés en noir. (Nouveau code.)
Lors de la planification de la transition du projet vers le sarcome, nous avons réalisé que nos experts en sarcome avaient des questions scientifiques sur le sarcome qu'ils voulaient explorer et qui nécessitaient plus de flexibilité dans la conception de l'unité de travail que ce qui était possible avec l'application existante. Ils voulaient explorer les différences entre deux ou plusieurs sous-types spécifiques de sarcome, et en exclure d'autres de l'analyse (voir figure 1D). (Par exemple, ils voulaient explorer des biomarqueurs qui distinguent les sous-types de LMS.) Malheureusement, la possibilité d'exclure des échantillons n'était pas intégrée à l'application originale Mapping Cancer Markers.
En travaillant ensemble, l'équipe Mapping Cancer Markers a conçu une petite extension à l'application qui ajouterait les capacités nécessaires, fournirait une flexibilité supplémentaire pour les besoins futurs et préserverait la compatibilité ascendante, tout en minimisant les changements de code totaux (Figure 1E).
Au cours des derniers mois, les bénévoles du World Community Grid ont traité des milliers d'unités de travail pour tester le nouveau code en version bêta. Ces changements nous donnent le pouvoir de procéder à des ajustements précis qui adaptent l'ensemble de données à la question précise que chaque unité de travail explorera.
Merci à tous ceux qui soutiennent Mapping Cancer Markers.
3 déc. 2020
traduction de l'article WCG : https://www.worldcommunitygrid.org/about_us/viewNewsArticle.do?articleId=669


