Voici une courte vidéo de David Baker décrivant certains résultats passionnants de la méthode "De novo design" ciblant le SRAS-Cov-2 (virus de la COVID-19)
Traduction de la vidéo : Mise-à-jour - coronavirus Rosetta@Home - Septembre 2020
Dans cette vidéo, le professeur David Baker décrit les progrès récemment réalisés dans la lutte contre le coronavirus. Avec l'aide de volontaires Rosetta@Home, des scientifiques de l'Institut pour la conception de protéines de l'Université de Washington ont réussi à créer des protéines antivirales candidates qui bloquent l'infection à coronavirus en laboratoire. Cette percée avance vers des essais cliniques.
"Merci à tous d'avoir contribué à Rosetta@Home : vous avez vraiment formé la colonne vertébrale de nos efforts informatiques de groupe. Je voulais partager avec vous, aujourd'hui, des résultats très intéressants sur la lutte contre le coronavirus avec la conception de protéines. L'une des choses que nous avons faite sur Rosetta@Home est de concevoir des séquences qui se replient en de nouvelles structures de forme complémentaire au virus et nous avons conçu des millions de ces structures et c'est pourquoi cela prend beaucoup de temps de calcul puis, après avoir trouvé notre forme complémentaire, nous concevons leurs surfaces afin qu'elles puissent se lier étroitement au virus. Nous avons fabriqué ces protéines en laboratoire et nous avons trouvé celles qui se lient très étroitement à la protéine virale, de façon plus fine que les protéines précédentes. Nous avons constaté, en outre, que ces protéines protègent les cellules du coronavirus vivant et nous sommes donc très excités à ce sujet comme thérapeutique potentielle et nous essayons de savoir comment nous pourrions avancer sa sortie mondiale. Rien de tout cela ne pourrait être vraiment fait sans vos contrubutions et je voulais vous remercier pour ça. Si vous souhaitez en savoir plus sur une partie de ce travail, nous avons publié un article scientifique, la semaine dernière que vous pouvez trouver sur notre site web (traduction ci-dessous). Il y a aussi eu une bonne quantité de choses dans les médias à ce sujet mais, de toute façon, je voulais vous remercier beaucoup pour vos contributions et nous allons de l'avant pour avoir une affinité de liants encore meilleure. Nous nous appuyons beaucoup sur Rosetta@Home alors j'espère que vous resterez impliqués sur ce projet."
David Baker, Ph.D.
Directeur de Rosetta@Home Professeur de Médecine (Institut pour la conception de protéines - Université de Washington)
Pour plus d'informations, veuillez consulter la publication : De novo design of picomolar SARS-CoV-2 mini protein inhibitors
(traduction ci-dessous)
Méthode "De novo design" des inhibiteurs de la miniprotéine picomolaire SARS-CoV-2
Extrait
Cibler l'interaction entre la protéine Spike du SRAS-CoV-2 et le récepteur ACE2 humain est une stratégie thérapeutique prometteuse. Nous avons conçu des inhibiteurs en utilisant deux approches de conception "de novo". Les échafaudages générés par ordinateur ont été construits autour d'une hélice ACE2 qui interagit avec le domaine de liaison au récepteur Spike (RBD), ou ancrés contre le RBD pour identifier de nouveaux modes de liaison et leurs séquences d'acides aminés conçues pour optimiser la liaison, le repliement et la stabilité de la cible. Dix conceptions ont lié le RBD avec des affinités allant de 100 pM à 10 nM, et ont bloqué l'infection ARS-CoV-2 des cellules Vero E6 avec des valeurs d'IC 50 entre 24 pM et 35 nM; Les plus puissants, avec de nouveaux modes de liaison, sont les protéines de 56 et 64 résidus (IC 50 ~ 0,16 ng / ml). Les structures de microscopie cryoélectronique de ces minibindres en complexe avec le trimère d'ectodomaine de pointe SARS-CoV-2 avec les trois RBD liés sont presque identiques aux modèles de calcul. Ces minibinders hyperstables fournissent des points de départ pour les thérapies contre le SRAS-CoV-2.
L'infection par le SRAS-CoV-2 débute généralement dans la cavité nasale, le virus s'y répliquant pendant plusieurs jours avant de se propager aux voies respiratoires inférieures. L'administration d'une concentration élevée d'un inhibiteur viral dans le nez et dans le système respiratoire pourrait donc généralement fournir une protection prophylactique et / ou un bénéfice thérapeutique pour le traitement d'une infection précoce, et pourrait être particulièrement utile pour les travailleurs de la santé et d'autres personnes entrant en contact fréquent avec des personnes infectées. Un certain nombre d'anticorps monoclonaux sont en cours de développement en tant que traitements systémiques du COVID-19, mais ces protéines ne sont pas idéales pour l'administration intranasale car les anticorps sont des molécules volumineuses et souvent pas extrêmement stables et la densité des sites de liaison est faible (deux pour 150 KDa d'anticorps); L'amélioration de la maladie dépendante des anticorps est également un problème potentiel. Les liants de protéines Spike de haute affinité qui bloquent l'interaction avec l'enzyme 2 (ACE2) de conversion de l'angiotensine du récepteur cellulaire humain avec une stabilité améliorée et des tailles plus petites pour maximiser la densité des domaines inhibiteurs pourraient avoir des avantages par rapport aux anticorps pour une administration directe dans le système respiratoire par administration intranasale, nébulisation ou aérosol de poudre sèche. Nous avons découvert précédemment que l'administration intranasale de petites protéines conçues pour se lier étroitement à l'hémagglutinine de la grippe peut fournir une protection à la fois prophylactique et thérapeutique dans des modèles de d'infection grippale mortelle.
Stratégie de conception
Nous avons entrepris de concevoir des minibinders de protéines de haute affinité pour le SARS-CoV-2 Spike RBD qui entrent en compétition avec la liaison ACE2. Nous avons exploré deux stratégies: d'abord, nous avons incorporé l'hélice alpha de ACE2 qui fait la majorité des interactions avec le RBD dans de petites protéines conçues qui font des interactions supplémentaires avec le RBD pour atteindre une affinité plus élevée ( Fig. 1A ). Deuxièmement, nous avons conçu des liants complètement à partir de zéro sans nous fier aux interactions de liaison RBD connues ( Fig.1B). Un avantage de la deuxième approche est que la gamme des possibilités de conception est beaucoup plus large, et donc potentiellement une plus grande diversité de modes de liaison à haute affinité peut être identifiée. Pour la première approche, nous avons utilisé le constructeur de plans Rosetta pour générer des miniprotéines qui incorporent l'hélice ACE2 (résidus ACE2 humains 23 à 46). Pour la deuxième approche, nous avons utilisé l'amarrage RIF ( 12 ) et la conception à l'aide de grandes bibliothèques de miniprotéines pour générer des liants vers des régions distinctes de la surface RBD entourant le site de liaison ACE2 ( Fig. 1 et Fig. S1).
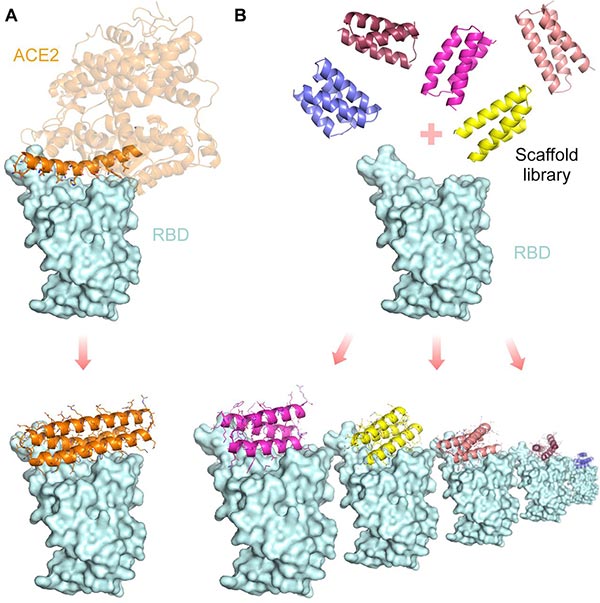
Fig. 1 Vue d'ensemble des approches de conception informatique.
( A ) Conception de protéines hélicoïdales incorporant l'hélice ACE2.
( B ) Conception de novo à grande échelle de petits échafaudages hélicoïdaux (en haut) suivi d'un amarrage de champ d'interaction rotamère (RIF) pour identifier la forme et les modes de liaison chimiquement complémentaires.
Caractérisation expérimentale et optimisation
De grands pools de minibinders conçus (voir Méthodes) réalisés en utilisant les première et seconde approches, ont été codés dans de longs oligonucléotides et criblés pour la liaison à RBD marqué par fluorescence affiché sur la surface des cellules de levure. Le séquençage en profondeur a identifié trois conceptions d'échafaudage en hélice ACE2 ( approche 1 ) et 105 conceptions d'interface de novo ( approche 2 ) qui ont été enrichies après le tri cellulaire activé par fluorescence (FACS) pour la liaison RBD. Les trois modèles d'échafaudage ACE2 et douze des modèles de novo ont été exprimés en E. coliet purifié. L'une des conceptions à échafaudage ACE2 et onze des douze conceptions de novo étaient RBD soluble et lié avec des affinités allant de 100 nM à 2 uM dans des expériences d'interférométrie biocouche (BLI) (figures S2, A, C et E et S3). La maturation d'affinité de la conception à échafaudage ACE2 par mutagenèse par PCR a conduit à un variant, AHB1, qui a lié RBD avec une affinité de ~ 1 nM (fig. modèle de conception, mais avait une faible thermostabilité (fig. S4C). Nous avons généré dix conceptions supplémentaires incorporant l'épingle à cheveux hélicoïdale de liaison de AHB1, et avons constaté que l'une liait le RBD et était thermostable (fig. S2, B, D et F).
Pour 50 des minibinders fabriqués à l'aide de l'approche 2 et de la conception à échafaudage en hélice ACE2 de deuxième génération, nous avons généré des bibliothèques de mutagenèse à saturation de site (SSM) dans lesquelles chaque résidu de chaque conception a été remplacé par chacun des 20 acides aminés un à la fois. Un séquençage en profondeur avant et après le tri FACS pour la liaison RBD a révélé que les résidus à l'interface de liaison et au noyau de la protéine étaient largement conservés pour 40 des 50 minibinders Approach 2 et pour la conception à échafaudage hélice ACE2 ( Fig.2et figues. S6 et S7). Pour la plupart de ces minibinders, un petit nombre de substitutions a été enrichi dans le tri FACS; Des bibliothèques combinatoires incorporant ces substitutions ont été construites pour la conception basée sur ACE2 et les huit conceptions d'Approche 2 à affinité la plus élevée et à nouveau criblées pour la liaison à la RBD à des concentrations allant jusqu'à 20 pM. Chaque bibliothèque a convergé sur un petit nombre de séquences étroitement liées; l'un de ceux-ci a été sélectionné pour chaque conception AHB2 ou LCB1-LCB8 et s'est avéré se lier au RBD avec une affinité élevée sur la surface de la levure d'une manière concurrencée par ACE2 ( Fig. 3 et Fig. S8).
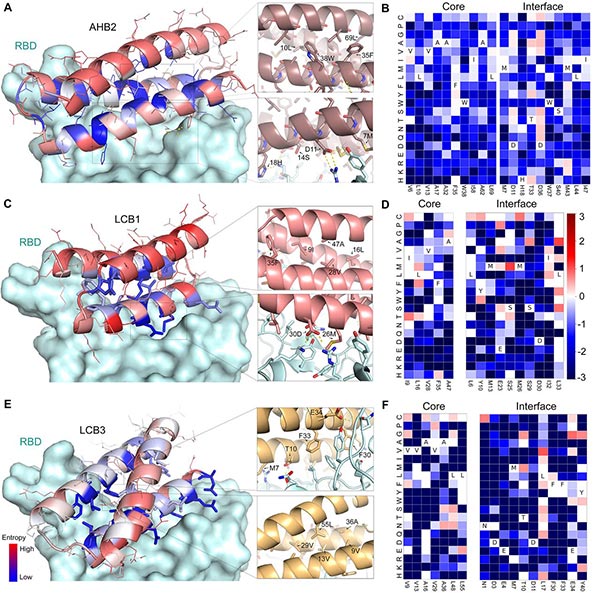
Fig. 2 Cartographie de séquence haute résolution de AHB2, LCB1 et LCB3 avant l'optimisation de la séquence.
( A , C et E ) Les protéines de liaison conçues sont colorées par l'entropie de position de Shannon de la mutagenèse de saturation du site avec des positions bleues indiquant une faible entropie (conservées) et en rouge celles de haute entropie (non conservées). ( B , D et F) Cartes thermiques représentant les valeurs d'enrichissement de liaison RBD pour des mutations uniques dans le noyau du modèle de conception (à gauche) et l'interface conçue (à droite). Les substitutions qui sont fortement épuisées sont indiquées en bleu et les mutations bénéfiques en rouge. L'épuisement de la plupart des substitutions à la fois dans le site de liaison et le noyau suggère que les modèles de conception sont largement corrects, tandis que les substitutions enrichies suggèrent des voies pour améliorer l'affinité. Les cartes SSM complètes sur toutes les positions pour AHB2 et les huit conceptions de novo sont fournies dans les fig. S6 et S7.
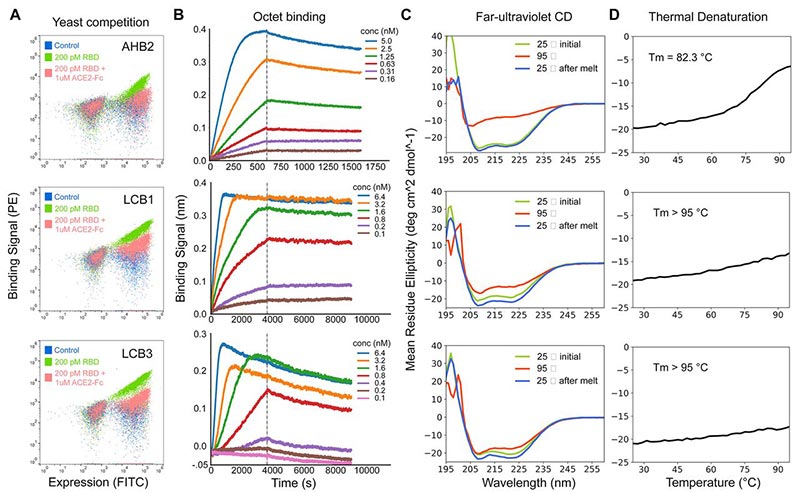
Fig. 3 Les conceptions optimisées se lient avec une forte affinité à la RBD, sont en concurrence avec ACE2 et sont thermostables.
( A ) ACE2 est en concurrence avec les conceptions pour la liaison à la RBD. Les cellules de levure présentant la conception indiquée ont été incubées avec 200 pM de RBD en présence ou en l'absence de 1 uM d'ACE2, et la liaison de RBD aux cellules (axe Y) a été surveillée par cytométrie en flux. ( B ) Liaison des miniprotéines purifiées à la RBD surveillée par interférométrie biocouche. Pour les LCB1 et LCB3, les K d n’ont pas pu être estimés avec précision en raison du manque de sensibilité de l’instrument et des longs temps d’équilibrage inférieurs à 200 pM. ( C ) Spectres de dichroïsme circulaire à différentes températures, et ( D ) signal CD à une longueur d'onde de 222 nm en fonction de la température. Les conceptions entièrement de novo LCB1 et LCB3 sont plus stables que la conception à hélice échafaudée ACE2 AHB2.
AHB2 et LCB1 – LCB8 ont été exprimés, purifiés à partir d' E. Coli et la liaison à la RBD évaluée par BLI. Pour sept des plans, les valeurs K D allaient de 1 à 20 nM ( Fig.3 , fig. S8 et tableau S2), et pour deux (LCB1 et LCB3), les valeurs K D étaient inférieures à 1 nM, ce qui est trop forte pour mesurer de manière fiable avec cette technique ( Fig. 3 ). A la surface des cellules de levure, LCB1 et LCB3 ont montré des signaux de liaison à 5 pM de RBD après un traitement par protéase (trypsine et chymotrypsine) (fig. S9). Les spectres de dichroïsme circulaire des minibindres purifiés étaient conformes aux modèles de conception, et les températures de fusion pour la plupart étaient supérieures à 90 ° C ( Fig.3et fig. S8). Les dessins ont conservé une activité de liaison complète après 14 jours à température ambiante (fi g.S10). AHB1 / 2 et LCB3 se sont également liés au SRAS-CoV RBD (en plus du SARS-CoV-2 RBD), mais avec une affinité plus faible (fig. S11); nous prévoyons que les affinités de liaison obtenues pour le SRAS-CoV-2 pourraient être facilement obtenues pour d'autres protéines de pointe de coronavirus si celles-ci étaient directement ciblées pour la conception.
Détermination de la structure CryoEM
Nous avons caractérisé les structures de LCB1 et LCB3 en complexe avec le trimère d'ectodomaine de pointe SARS-CoV-2 à une résolution de 2,7 Å et 3,1 Å, respectivement, et avons constaté que les minibinders se lient de manière stoechiométrique aux trois RBD dans le trimère de pointe ( Fig.4, A et E , et figures S12 et S13). Bien que le pic abritait principalement deux RBD ouverts pour les deux complexes, nous avons identifié un sous-ensemble de particules avec trois RBD ouverts pour le complexe LCB3 ( Fig. 4, A et E , et figures S12 et S13). Nous avons amélioré la résolvabilité des densités RBD / LCB1 et RBD / LCB3 en utilisant une classification ciblée et un raffinement local donnant des cartes à une résolution de 3,1 et 3,5 Å permettant la visualisation des interactions formées par chaque minibinder avec le RBD ( Fig.4, B et F , et figures S12 et S13).
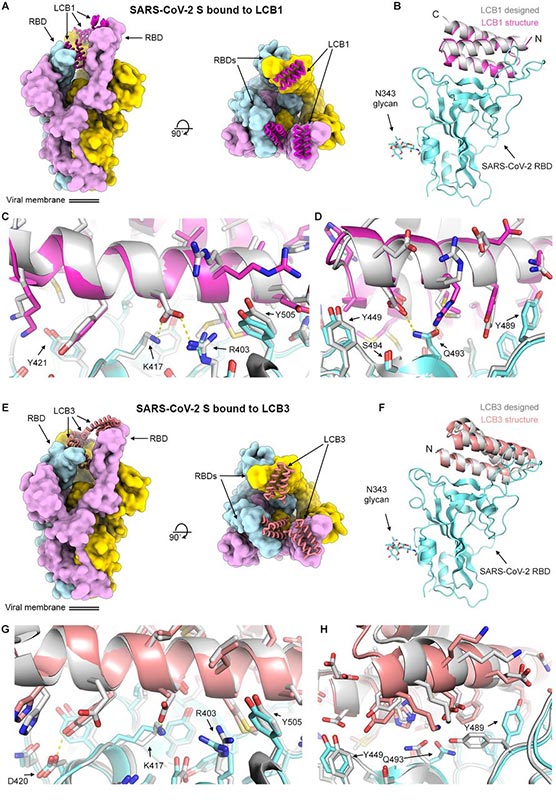
Fig.4 Caractérisation CryoEM des minibindres LCB1 et LCB3 en complexe avec SARS-CoV-2 S.
( A ) Représentation de surface moléculaire de LCB1 lié au trimère de l'ectodomaine SARS-CoV-2 S vu le long de deux orientations orthogonales.
( B ) Superposition du modèle conçu (gris argent) et de la structure cryoEM raffinée (magenta) de LCB1 (en utilisant la carte obtenue par raffinement local) lié au RBD (cyan).
( C et D ) Vues agrandies du modèle de calcul (gris argent) du complexe LCB1 / RBD superposé sur la structure CryoEM (cyan pour RBD et rose pour LCB1) montrant des chaînes latérales en interaction sélectionnées.
( E ) Représentation de surface moléculaire de LCB3 lié au trimère de l'ectodomaine SARS-CoV-2 S vu le long de deux orientations orthogonales.
( F) Superposition du modèle conçu (gris argent) et de la structure cryoEM raffinée (saumon) de LCB3 (en utilisant la carte obtenue par raffinement local) lié au RBD (cyan).
( G et H ) Zoom sur les interactions entre LCB3 (saumon) et le SRAS-CoV-2 RBD (cyan) montrant des chaînes latérales d'interaction sélectionnées.
En (A) et (E), chaque protomère S est coloré distinctement (cyan, rose et or).
Pour (B) et (F), les RBD ont été superposés pour évaluer les écarts de pose de liaison entre les modèles conçus et la structure raffinée de chaque minibinder.
LCB1 et LCB3 accostent avec des orientations opposées dans la crevasse formée par le motif de liaison au récepteur RBD grâce à des interfaces complémentaires de forme étendue avec de nombreuses interactions électrostatiques médiées par deux des trois hélices three minibinder ( Fig.4, B à D et F à H ). Semblable à ACE2, les sites de liaison LCB1 et LCB3 sont enterrés dans l'état conformationnel S fermé et nécessitent l'ouverture d'au moins deux RBD pour permettre la reconnaissance simultanée des trois sites de liaison ( Fig. 4, A et E ). LCB1 et LCB3 forment des liaisons hydrogène multiples et des ponts salins avec le RBD avec des surfaces enterrées de ~ 1000Å ^ 2 et ~ 800Å ^ 2, respectivement ( Fig.4, C, D, G et H), compatible avec les affinités sous-nanomolaires de ces inhibiteurs. Tel que conçu, les sites de liaison pour LCB1 et LCB3 chevauchent celui de ACE2 (fig. S14 et tableau S1), et devraient donc entrer en compétition pour la liaison à la RBD et inhiber l'attachement viral à la surface de la cellule hôte.
La superposition des modèles LCB1 / RBD ou LCB3 / RBD aux structures cryoEM correspondantes, en utilisant le RBD comme référence, montre que les poses de liaison correspondent étroitement à la conception avec le squelette Cɑ rmsd de 1,27 Å et 1,9Å pour LCB1 et LCB3, respectivement ( Fig. 4, B et F ) et la plupart des interactions polaires dans les modèles de conception correspondent étroitement à la structure CryoEM ( Fig. 4, C, D, G et H ). Ces données montrent que la méthode de conception informatique peut avoir une précision assez élevée. Les comparaisons de structure sur la figure 4, C, D, G et H , concernent les modèles de conception d'origine; les substitutions qui augmentent l'affinité de liaison sont assez subtiles et ont très peu d'effet sur la géométrie du squelette.
Neutralisation virale
Nous avons étudié la capacité de AHB1, AHB2 et LCB1-5 à prévenir l'infection des cellules par le SRAS-CoV-2 de bonne foi . Des concentrations variables de minibinders ont été incubées avec 100 unités de formation de focalisation (FFU) de SARS-CoV-2, puis ajoutées à des monocouches Vero E6. AHB1 et AHB2 fortement neutralisés SRAS-CoV-2 (IC 50 de 35 nM et 15,5 nM , respectivement), tandis qu'un minibinder de la grippe de contrôle n'a montré aucune activité de neutralisation ( Fig. 5A ). Ensuite, nous avons testé les minibinders LCB1-5 conçus par Approach 2. Nous avons observé une neutralisation encore plus puissante du SARS-CoV-2 par LCB1 et LCB3 avec des valeurs IC 50 de 23,54 pM et 48,1 pM, respectivement ( Fig.5B; à des volumes d'incubation accrus, des CI50 aussi faibles que 11 pM ont été obtenues). Sur une base molaire, ces valeurs sont environ 3 fois inférieures à celles de l'anticorps monoclonal anti-SARS-CoV-2 le plus puissant décrit à ce jour ; sur une base de masse, en raison de leur très petite taille, les dessins sont encore plus puissants que les anticorps.
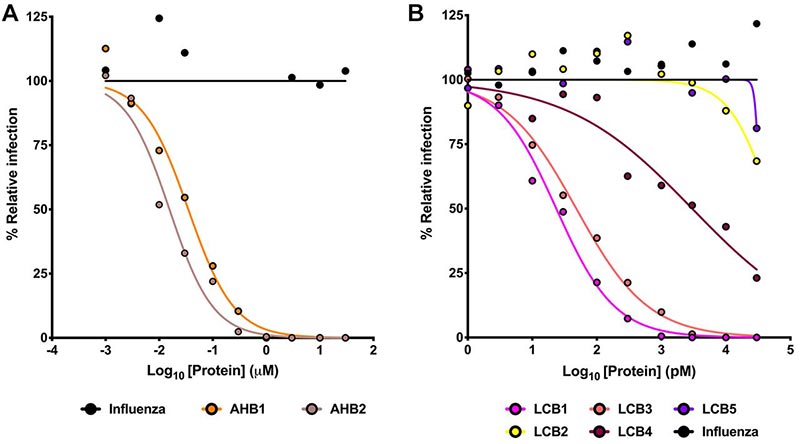
Fig. 5 Neutralisation du virus vivant par des inhibiteurs de miniprotéines conçus.
L'activité de neutralisation de ( A ) AHB1 et AHB2 ou ( B ) LCB1-5 a été mesurée par FRNT. Les concentrations indiquées de minibinders ont été incubées avec 100 FFU de SRAS-CoV-2 authentique et ensuite transférées sur des monocouches Vero E6. AHB1, AHB2, LCB1 et LCB3 neutralisent efficacement le SARS-CoV-2, avec des valeurs EC 50 <50nM (AHB1 et AHB2) ou <50pM (LCB1 et LCB3). Les données sont représentatives de deux expériences indépendantes, chacune réalisée en double technique.
Conclusions
Les minibinders conçus dans ce travail présentent des avantages potentiels par rapport aux anticorps en tant que thérapeutiques potentielles. Ensemble, ils couvrent une gamme de modes de liaison, et en combinaison, une fuite mutationnelle virale serait tout à fait improbable (figures S1 et S14 et tableau S1). Le maintien de l'activité après une période prolongée à des températures élevées suggère qu'ils ne nécessiteraient pas une chaîne d'approvisionnement à température contrôlée. Les conceptions sont 20 fois plus petites qu'une molécule d'anticorps complète et, par conséquent, à masse égale, ont 20 fois plus de sites de neutralisation potentiels, augmentant l'efficacité potentielle d'un médicament administré localement. Le coût des produits et la capacité de passer à une production très élevée devraient être inférieurs pour les miniprotéines beaucoup plus simples, qui, contrairement aux anticorps, ne nécessitent pas d'expression dans les cellules de mammifères pour un repliement correct. La petite taille et la stabilité élevée devraient également les rendre aptes à être formulés dans un gel pour application nasale et à être administrés directement dans le système respiratoire par nébulisation ou sous forme de poudre sèche. Nous explorerons d'autres voies de livraison dans les mois à venir alors que nous cherchons à traduire les protéines neutralisantes de haute puissance en traitements et prophylactiques contre le SRAS-Cov2. L'immunogénicité est un problème potentiel avec toute molécule étrangère, mais pour les petites protéines conçues de novo précédemment caractérisées, peu ou pas de réponse immunitaire a été observée ( Nous explorerons d'autres voies de livraison dans les mois à venir alors que nous cherchons à traduire les protéines neutralisantes de haute puissance en traitements et prophylactiques contre le SRAS-Cov2. L'immunogénicité est un problème potentiel avec toute molécule étrangère, mais pour les petites protéines conçues de novo précédemment caractérisées, peu ou pas de réponse immunitaire a été observée ( Nous explorerons d'autres voies de livraison dans les mois à venir alors que nous cherchons à traduire les protéines neutralisantes de haute puissance en traitements et prophylactiques contre le SRAS-Cov2. L'immunogénicité est un problème potentiel avec toute molécule étrangère, mais pour les petites protéines conçues de novo précédemment caractérisées, peu ou pas de réponse immunitaire a été observée (11 , 14 ), peut-être parce que la solubilité et la stabilité élevées associées à la petite taille rendent la présentation sur des cellules dendritiques moins probable.
Le moment choisi est essentiel en cas d'épidémie de pandémie: des traitements efficaces sont nécessaires dans les plus brefs délais. Nous avons commencé à concevoir minibinders en Janvier 2020 grâce à la Rosetta sur un modèle de la structure de Spike SRAS-CoV-2 et passe au structures cristallines une fois qu'ils sont devenus disponibles ( 4 , 15 - 17). À la fin du mois de mai 2020, nous avions identifié des neutralisants très puissants du virus infectieux; pendant ce même temps, un certain nombre d'anticorps monoclonaux neutralisants ont été identifiés. Nous pensons qu'avec un développement continu, l'approche de conception informatique peut devenir beaucoup plus rapide. Premièrement, à mesure que les méthodes de prédiction de la structure continuent de gagner en précision, des modèles cibles adaptés à la conception pourraient être générés dans la journée suivant la détermination de la séquence du génome d'un nouveau pathogène. Deuxièmement, avec l'amélioration continue des méthodes de conception informatique, il devrait être possible de rationaliser le flux de travail décrit ici, qui nécessitait le criblage de grands ensembles de conceptions informatiques, suivi d'une optimisation expérimentale, pour identifier les liants à très haute affinité. L'accord très étroit des structures cryoEM de LCB1 et LCB3 avec les modèles de conception informatique suggère que les principaux défis à surmonter ne sont pas dans la conception de novo de protéines avec une forme et une complémentarité chimique à la surface cible, mais dans la reconnaissance des meilleurs candidats et identifier un petit nombre de substitutions augmentant l'affinité. La grande quantité de données collectées dans les expériences de conception d'interface de protéines telles que celles décrites ici devrait informer l'amélioration des modèles atomiques détaillés au cœur des calculs de conception de Rosetta, ainsi que des approches d'apprentissage automatique complémentaires, pour permettre la reconnaissance et l'amélioration de la conception de séquence du meilleurs candidats; cela permettrait une conception in silico encore plus rapide des inhibiteurs de pM comme LCB1 et LCB3. Avec le développement continu des méthodes, nous pensons qu'il deviendra possible de générer des modèles neutralisant les pathogènes à ultra haute affinité dans les semaines suivant l'obtention de la séquence du génome. Il est difficile de se préparer à de futures pandémies inconnues et une telle capacité pourrait être un élément important d’une stratégie d’intervention générale.
traduction :
- forum Rosetta : https://boinc.bakerlab.org/rosetta/forum_thread.php?id=14226
- article Science Mag : https://science.sciencemag.org/content/early/2020/09/08/science.abd9909


