
L'équipe OpenZika a publié un nouveau document de recherche décrivant les progrès qu'elle a réalisés après son partenariat avec WCG
Projet : OpenZika
Published on: 9 févr. 2023
Bref historique du projet OpenZika
Le Dr Carolina Horta Andrade (Faculdade de Farmácia Universidade Federal de Goiás, LabMo ), avec le Dr Sean Ekins ( Collaborations Pharmaceuticals, Inc. ) et le Dr Alexander Perryman (Rutgers University–New Jersey Medical School) ont lancé le projet OpenZika en mai . de 2016 en collaboration avec IBM WCG pour identifier d'éventuels inhibiteurs du virus Zika. Les patients touchés par ce virus souffrent de paralysie de leur système nerveux et les enfants nés de mères touchées par le virus présentent de graves troubles du développement cérébral. Alors qu'il constituait déjà une menace énorme au Brésil, il avait le potentiel de devenir une menace mondiale à moins qu'un traitement ne soit développé rapidement. La puissance de calcul massive de WCG a permis à l'équipe de tester rapidement des millions de composés à la recherche d'inhibiteurs possibles de diverses cibles du virus Zika.
En novembre , la liste des 7 600 composés potentiels a été passée au crible pour tester leur efficacité contre l'hélicase NS3, une protéine du virus Zika qui lui permet de dérouler son ARN duplex. La liste des molécules a été réduite à huit composés, dont cinq ont ensuite été vérifiés comme étant sûrs par une étude menée à l'Université de Californie à San Diego. En mars 2017 , l'équipe était prête à passer à la deuxième étape du projet. L'équipe a utilisé un serveur pour créer une grande bibliothèque de 30,2 millions de composés à tester contre la protéase NS2B-NS3, l'hélicase NS3 et les protéines NS5-polymérase. L'exécution d'un criblage informatique sur cette grande bibliothèque n'a été possible que grâce aux efforts combinés des bénévoles du World Community Grid (WCG).
Les tests sur ces 30,2 millions de composés se sont poursuivis jusqu'en décembre 2018 , lorsque l'équipe a analysé une base de données de composés supplémentaires fournie par ChemBridge. La liste d'un million de composés a été testée dans son efficacité contre la polymérase NS5, la méthyltransférase NS5 et l'hélicase NS3 ; le réduisant finalement à 55 composés d'intérêt. Les résultats ont été envoyés à l'Université de Californie pour évaluation dans le virus, ainsi qu'à l'Université de Sao Paulo pour évaluation avec les protéines du virus Zika en juillet 2019 .
Propulsée par une communauté de 80 000 volontaires qui ont fait don de près de 73 années CPU par jour en moyenne au projet WCG, l'équipe OpenZika a pu finaliser sa modélisation et le projet s'est achevé en décembre 2019. La phase suivante s'est concentrée sur la validation expérimentale et priorisation des molécules sélectionnées.
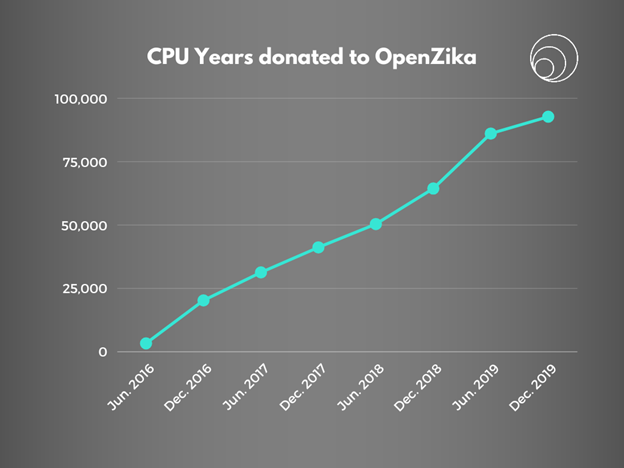
Figure 1 : Le nombre total d'années CPU données au projet OpenZika a augmenté régulièrement de mai 2016 à décembre 2019.
Nouveaux développements
En octobre 2022, les quatre années de recherche de l'équipe ont abouti à une publication dans le Journal of Chemical Information and Modeling[1].
L'article met en évidence le processus de calcul et les étapes de validation effectuées pendant et après l'analyse WCG. L'équipe de recherche a publié les résultats pour trois protéines importantes du virus Zika : NS3hel, NS2B-NS3pro et NS5 RdRp. À l'aide de WCG, les chercheurs ont recherché des millions de composés disponibles dans le commerce et identifié 61 composés d'intérêt possibles pour un criblage et une optimisation plus poussés.
Après les calculs d'amarrage massifs, les composés ont été filtrés à l'aide de modèles d'apprentissage automatique développés par le groupe LabMol pour identifier ceux qui étaient cytoprotecteurs contre l'infection par le virus Zika. Ensuite, les composés dont on prévoyait qu'ils passeraient la barrière hémato-encéphalique ont été retenus, afin de sélectionner ceux qui pourraient contrecarrer les effets du virus sur le système nerveux central. Enfin, une inspection de chimie médicinale a identifié et sélectionné des composés présentant des caractéristiques souhaitables présents dans les médicaments existants.
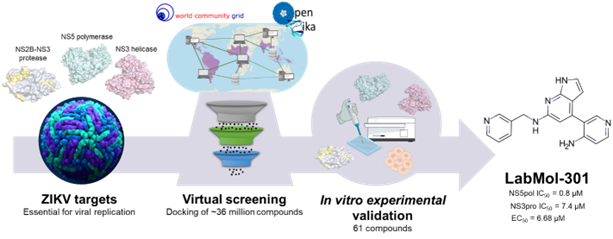
Figure 2 : Description du pipeline de découverte ; créé par Bruna KP Sousa du laboratoire du Dr C. Horta Andrade
Sur près de 404 millions de résultats générés par WCG, représentant près de 93 000 ans de calcul, 61 résultats ont été priorisés pour les tests. À l'aide d'essais enzymatiques et phénotypiques, cinq composés ont finalement été sélectionnés car ils inhibaient la fonction ou déstabilisaient les trois protéines virales d'intérêt, la protéase NS2B-NS3, l'hélicase NS3 et les protéines NS5-polymérase. D'autres tests ont identifié 8 composés comme étant capables de protéger les cellules de la mort causée par le virus, tout en montrant une faible toxicité cellulaire dans les cellules dérivées du foie et des reins. Les deux ensembles de 5 et 8 molécules se chevauchent pour deux composés, nommés par les auteurs LabMol-301 et LabMol-212.
Dr Carolina Horta Andrade : « Ces travaux ont démontré l'importance de l'intégration des approches informatiques et expérimentales, ainsi que le potentiel des réseaux collaboratifs à grande échelle pour faire avancer les projets de découverte de médicaments pour les maladies négligées et les virus émergents, malgré le manque de ressources disponibles. données sur l'activité antivirale directe et l'effet cytoprotecteur, qui reflètent l'affirmation des prédictions informatiques.
Les résultats de cette recherche sont passionnants et une optimisation plus poussée pourrait conduire à tester ces molécules en tant que traitements antiviraux du virus Zika. Les chercheurs recherchent maintenant des partenaires pour effectuer l'optimisation hit-to-lead avec synthèse chimique et autres validations expérimentales.
Nous remercions les bénévoles qui ont rendu ces découvertes possibles, ainsi que l'équipe OpenZika pour avoir partagé cette mise à jour passionnante et leur implication continue avec WCG. Si vous avez des commentaires ou des questions, veuillez les laisser dans ce fil pour que nous puissions y répondre. Merci pour votre soutien.
Traduction : Kalianthys
Source : https://www.worldcommunitygrid.org/about_us/article.s?articleId=779


